VERA PANCALDI
Descriptions multi-omiques du microenvironnement tumoral
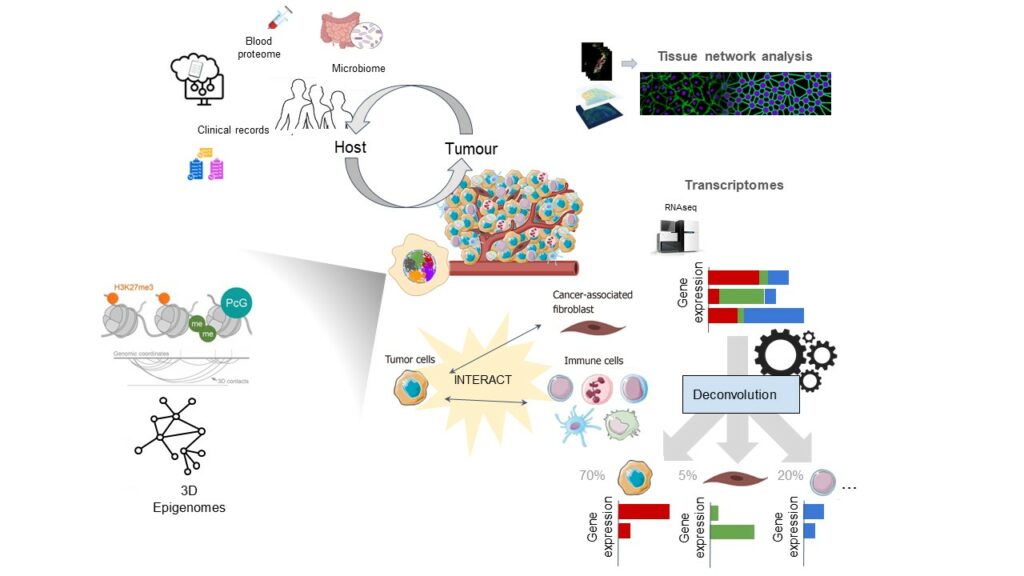
La première étape vers une meilleure compréhension du TME pour espérer un jour le contrôler, consiste à le décrire avec précision. Notre objectif est d’exploiter des ensembles de données cliniques de grandes cohortes en combinaison avec une caractérisation plus approfondie des sous-ensembles d’échantillons plus petits pour extraire autant d’informations spécifiques au patient que possible et initialiser les modèles de MET.
Cela implique 3 approches complémentaires :
- Quantification des proportions de type et d’état de cellules par déconvolution d’ensembles de données RNAseq en bulk.
Nous avons développé des outils pour quantifier et décrire les populations cellulaires dans le MET en utilisant plusieurs types de déconvolution. Dans la déconvolution basée sur la référence, nous utilisons des profils moléculaires de référence pour des types de cellules spécifiques et des données en bulk provenant des échantillons de tumeurs que nous combinons à l’aide d’approches statistiques pour estimer la proportion de cellules de chaque type présentes dans le mélange.
GEMDeCan - Quantification des empreintes spatiales des cellules dans les tissus tumoraux par analyse d’ensembles de données omiques spatiales
Tysserand et MOSNA
Notre équipe développe des outils pour analyser les données hautement complexes générées par ces méthodes, et nous pensons que les réseaux fournissent une méthode puissante pour analyser les expériences omiques spatiales. La bibliothèque tysserand peut reconstruire des réseaux spatiaux à partir d’expériences omiques résolues spatialement, et la bibliothèque mosna (Multi Omics Spatial Networks Analysis) fournit des méthodes pour analyser ces réseaux spatiaux afin d’étudier les interactions cellule-cellule et de trouver des composantes cellulaires voisines pouvant avoir un intérêt clinique potentiel. - Étude des épigénomes 3D des cellules cancéreuses et autres cellules dans le MET (cellules immunitaires, fibroblastes associés au cancer) pour mettre en évidence le couplage entre le contexte tissulaire, les paysages épigénomiques et les états de différenciation.
Nous avons récemment développé 2 outils pour la communauté scientifique intéressée par l’intégration de données épigénomiques dans un contexte de structure de la chromatine : GARDEN-NET est un navigateur de génome 3D accessible via une interface web, ChAseR est un package R facilitant l’intégration d’ensembles de données épigénomiques avec des réseaux de contact de chromatine et le calcul de l’assortativité, qui estime une association entre une caractéristique donnée (également définie par l’utilisateur) et des interactions 3D.
Nous avons appliqué ces outils à l’étude des principes d’organisation du génome en analysant l’expression des gènes, la méthylation de l’ADN, les modifications des histones, les origines de réplication de l’ADN et le moment de la réplication ainsi que récemment l’âge des gènes.
See version in English for papers and grants